KCSG 소식

- 알림
- KCSG 소식
| 주요 연구성과 | 간행물 | 미디어 | 연구회동정 | KCSG 인사이트 | 뉴스레터 |
[KCSG BR18-14] Sci Rep. 2023 Jun 19;13(1):9928.
- 작성자 관리자
- 등록일 2023-12-06
- 조회수 908
첨부파일
▶ KCSG BR18-14
Genomic analysis of plasma circulating tumor DNA in patients with heavily pretreated HER2 + metastatic breast cancer
제1저자 이경민(고대구병원)
Q. 연구의 배경은?
Trastuzumab을 위시한 여러 HER2 targeted agent의 도입으로 HER2 양성 유방암 환자들의 예후는 이전에 비해 매우 향상되었습니다. 그러나 아무리 좋은 약제라도 필연적으로 약제 내성이 발생하게 됩니다. 특히 여러 라인의 치료가 거듭될수록 다양한 내성 기전이 축적되게 되며, tumor clonal evolution으로 원발암과는 전혀 다른 분자유전학적 특징을 보이기도 합니다. 그러나 이를 파악하기 위해 특정 치료 시점마다 종양 생검을 진행하는 것은 매우 어렵고, 한 군데의 조직획득 만으로는 intra- or inter-tumor heterogeneity로 인해 부분적인 결과만 얻을 수도 있습니다.
Q. 연구의 목적은?
본 연구에서는 이전 여러 차례 치료를 받은 heavily treated HER2 breast cancer 환자들의 plasma ctDNA를 분석하여 주요 내성 변이를 확인하고 치료 및 예후 예측 인자를 탐색하고자 하였습니다.
Q.연구 방법?
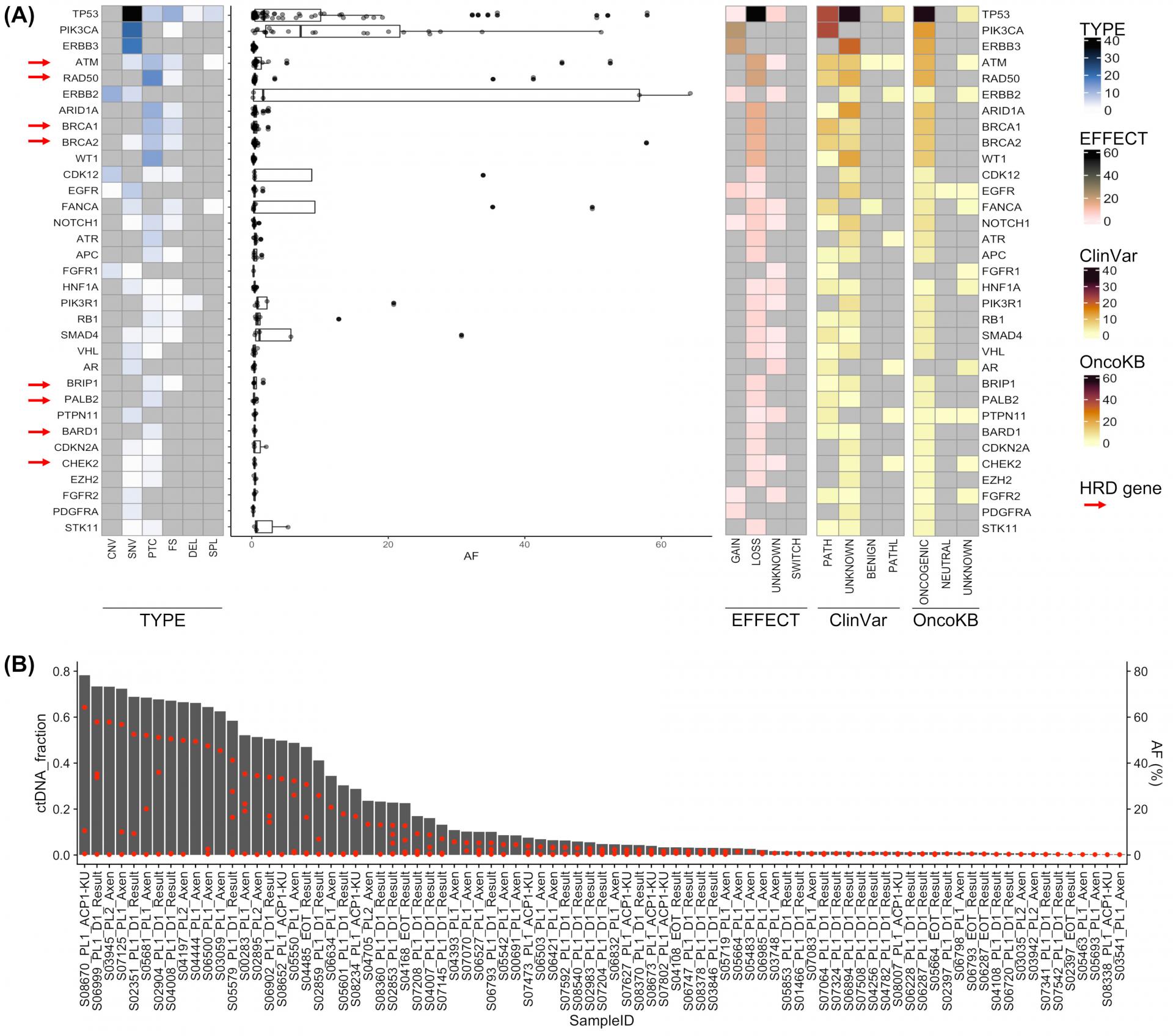
본 연구는 KCSG BR18-14/KM10B 연구에 참여했던 환자들에게서 수집한 혈액 샘플을 이용하여 수행되었습니다. Plasma ctDNA 시퀀싱은 K-MASTER project의 일환으로 K-Master Axen™ Cancer Panel을 사용하여 진행되었고, 각 변이의 pathogenicity는 ClinVar와 OncoKB 데이터베이스를 사용하여 분석하였습니다.
Q. 연구 결과?
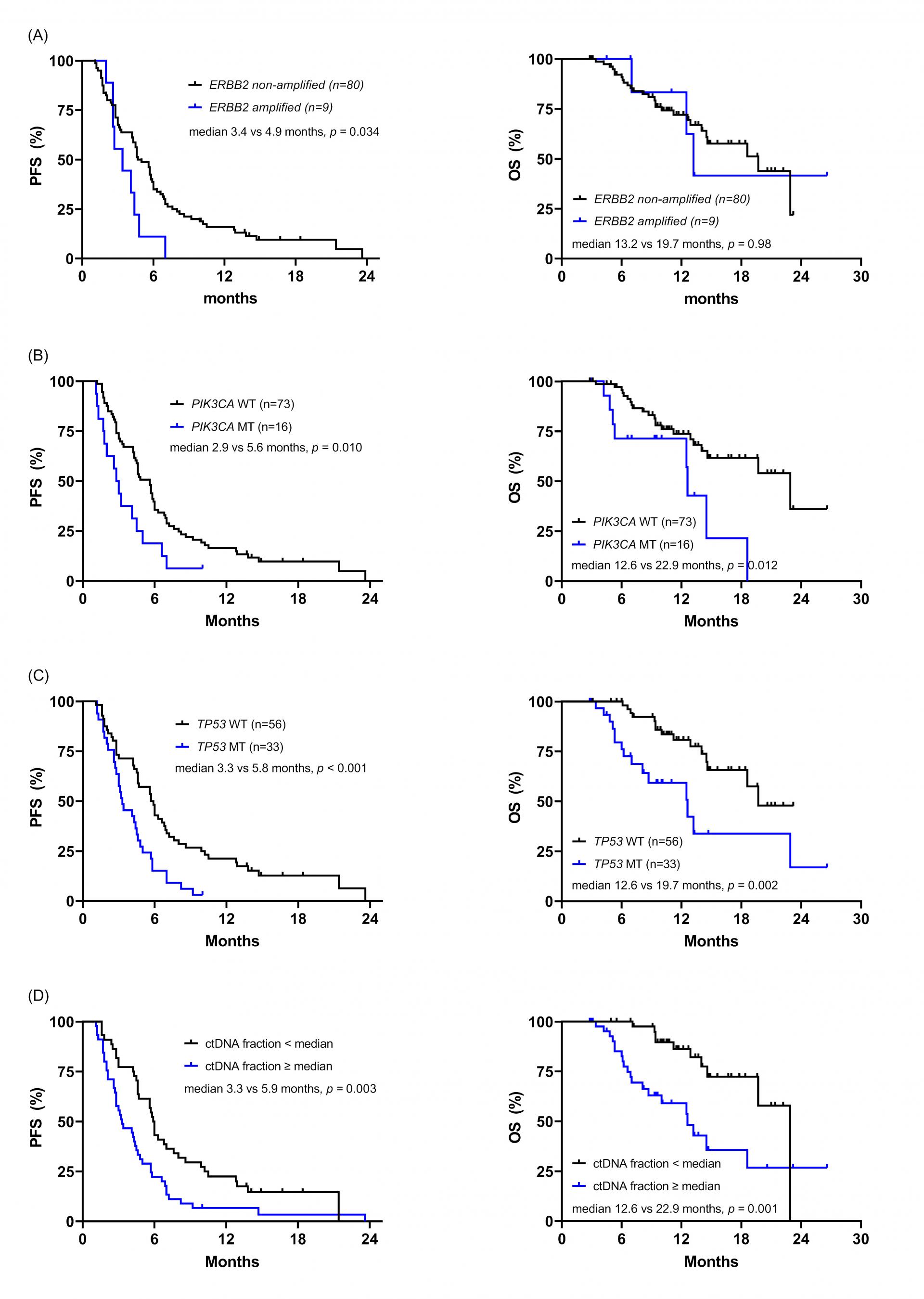
총 93명의 환자로부터 얻은 99개의 혈장 샘플을 분석에 활용했습니다. 이 중 92개는 임상 연구 참여 시기에 수집되었으며, 나머지 7개는 치료 종료(EOT) 시점에 추가로 획득된 샘플이었습니다. 분석 결과 TP53, PIK3CA, 그리고 ERBB3 유전자의 변이가 높은 빈도로 나타났는데, 특히 TP53와 PIK3CA 유전자 변이가 ctDNA에서 검출된 환자들의 생존율이 유의하게 낮았습니다. 또 ctDNA 분율(ctDNA fraction)을 계산하였을 때, ctDNA 분율이 높은 환자들의 예후가 좋지 않았습니다. 더불어 진단시점의 조직에서보다 ctDNA 분석에서 HRD 관련 유전자 변이가 더 높은 빈도로 나타났으며, 이를 가진 환자들의 예후 또한 좋지 않아 여러 항암 치료가 지속되며 HRD 관련 유전자 변이가 축적되는 것이 내성 획득의 한 기전일 수 있음을 제시하였습니다.
[Figure1]
[Figure4]
Q. 마지막으로 하고 싶은 말은?
본 연구의 전반적인 유전체 분석을 도맡아 진행해 주신 고려대학교 의과대학 이종원 박사님과 최정민 교수님, 그리고 훌륭한 연구로 모아진 데이터를 분석할 수 있게 기회를 주시고 지도하여 주신 고려대학교 구로병원 박인혜 교수님께 깊은 감사를 드립니다. 본 연구는 대한항암요법연구회를 통해 진행되었던 KCSG BR18-14/KM10B 연구의 탐색적 생체 표지자 연구 (exploratory biomarker analysis)로 원 연구가 성공적으로 진행될 수 있게 도움 주신 여러 기관의 연구자 선생님들께 지면을 빌어 다시 한번 감사 인사 드립니다.
-
다음 글
-
이전 글
 회원검색
회원검색 통합검색
통합검색 교육신청
교육신청 연구검색
연구검색 데이터
데이터 TOP
TOP


